KAIST(총장 신성철)는 생명화학공학과 이상엽 특훈교수와 미국 캘리포니아대 샌디에이고캠퍼스(UCSD) 생명공학과 버나드 팔슨(Bernhard Palsson) 교수 공동연구팀이 인공지능을 이용해 단백질 서열로부터 전사인자를 예측하는 시스템인 `딥티팩터(DeepTFactor)'를 개발했다고 29일 밝혔다.
전사인자 (transcription factor)는 특정 DNA 서열에 특이적으로 결합해 유전 정보를 복사하는 과정인 유전자의 전사를 조절하는 단백질이다.
유기체의 전사인자를 찾는 것은 유기체의 전사 조절 시스템 분석을 위한 첫 단계다. 지금까지 새로운 전사인자를 찾기 위해서는 이미 알려진 전사인자와의 유사한 성질을 분석하거나, 머신러닝과 같은 데이터 기반 접근 방식을 이용했다.
하지만, 심층 학습을 이용한 예측 시스템의 경우 시스템 내부의 복잡한 연산 때문에 추론 과정을 직접 확인할 수 없는 `블랙박스(black box)'라는 특징을 가지고 있다.
심층 학습(딥러닝)은 문제 해결을 위한 잠재적인 특징을 내재적으로 학습할 수 있기에 최근 다양한 생물학 분야에서 활용되고 있다.
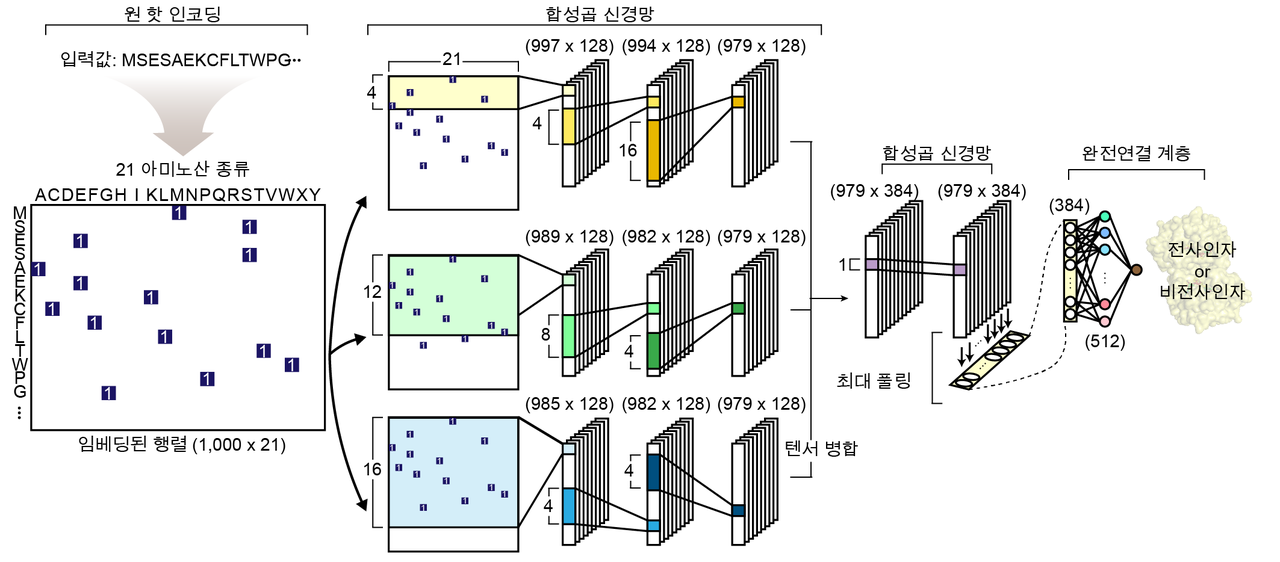
공동연구팀은 심층 학습 기법을 이용해 주어진 단백질 서열이 전사인자인지 예측할 수 있는 시스템인 딥티팩터(DeepTFactor)를 개발했다.
딥티팩터는 단백질 서열로부터 전사인자를 예측하기 위해 세 개의 병렬적인 합성곱 신경망(convolutional neural network)을 이용한다.
공동연구팀은 딥티팩터를 이용해 대장균의 전사인자 332개를 예측했으며, 그 중 3개의 전사인자의 게놈 전체 결합 위치(genome-wide binding site)를 실험으로 확인함으로써 딥티팩터의 성능을 검증했다.
공동연구팀은 나아가 딥티팩터의 추론 과정을 이해하기 위해 특징 지도 (saliency map) 기반의 심층 학습 모델 해석 방법론을 사용했다. 이를 통해 딥티팩터의 학습 과정에서 전사인자의 DNA의 결합 영역에 대한 정보가 명시적으로 주어지지 않았지만, 내재적으로 이를 학습해 예측에 활용한다는 사실을 확인했다.
연구팀에 따르면, 특정 생물군의 단백질 서열만을 위해 개발됐던 이전 예측 방법론들과 달리, 딥티팩터는 모든 생물군의 단백질 서열에서 우수한 성능을 보여 다양한 유기체의 전사 시스템 분석에 활용 가능할 것으로 기대된다.
이상엽 특훈교수는 “이번 연구에서 개발한 딥티팩터를 이용해서 새롭게 발견되는 단백질 서열과 아직 특성화되지 않은 수많은 단백질 서열을 높은 처리 능력으로 분석할 수 있게 됐다”며 “이는 유기체의 전자 조절 네트워크 분석을 위한 기초 기술로써 활용 가능할 것”이라고 밝혔다.
이번 공동연구의 저자는 김기배 박사과정 연구원(제1 저자), 예 가오(Ye Gao) (UCSD, 제2 저자)교수, 버나드 팔슨(Bernhard Palsson) 교수(UCSD, 제3 저자), 이상엽(교신저자) 교수 등 4명이다.
이번 연구는 국제학술지인 `미국국립과학원회보(PNAS)'에 12월 28일자로 게재됐다.
AI타임스 김재호 기자 jhk6047@aitimes.com