인공지능(AI)의 자기지도학습 방법론을 통해 적은 유전자 정보만으로 각 단일세포의 보편적인 특징부터 세부적인 특징까지 파악할 수 있는 기술 ‘sc로버스트(ScRobust)’가 개발됐다.
기존 방법론은 각 조직 별로 유전자 분포를 파악했다면, sc로버스트는 AI를 적용해 동일한 세포 종류라도 당뇨병의 정도에 따라 구분되는 세부적인 특징을 구별하고 심층분석에 활용할 수 있다.
광주과학기술원(GIST, 총장 임기철)은 AI대학원 이현주 교수 연구팀이 단일세포 RNA 시퀀싱 기술의 근본적인 한계를 극복할 수 있는 자기지도학습 방법론을 개발했다고 밝혔다.
자기지도학습은 별도의 라벨 없이 AI모델을 학습시키는 것으로, 이번 연구에는 자기지도학습 방법론 중 하나인 대조학습이 이용됐다. 대조 학습은 대상들의 차이를 더 명확하게 보여줄 수 있도록 학습하는 방법을 말한다.
여러 세포의 RNA가 섞인 유전자 발현량을 측정할 수 있는 다세포 RNA 시퀀싱 기술에 반해 단일세포 RNA 시퀀싱은 단일세포만을 대상으로 하기 때문에 측정 정확도가 떨어진다. 전체 유전자 중 10%만이 측정 가능한 높은 해상도를 가지고, 나머지 90% 정보는 낮은 해상도로 인하여 측정이 불가하다. 기존의 연구는 주로 여러 세포에서 공통으로 발현되는 약 10%의 유전자만을 사용하여 세포 유형을 예측하고 분석해 왔다.
하지만 특정 세포 종류에서만 발현되는 유전자가 오히려 해당 세포를 더 자세히 설명하는 경우가 많고, 현재 사용되는 단일세포 RNA 시퀀싱 기술은 약 90%의 유전자 정보를 사용하지 못하는 근본적인 문제점이 있었다.
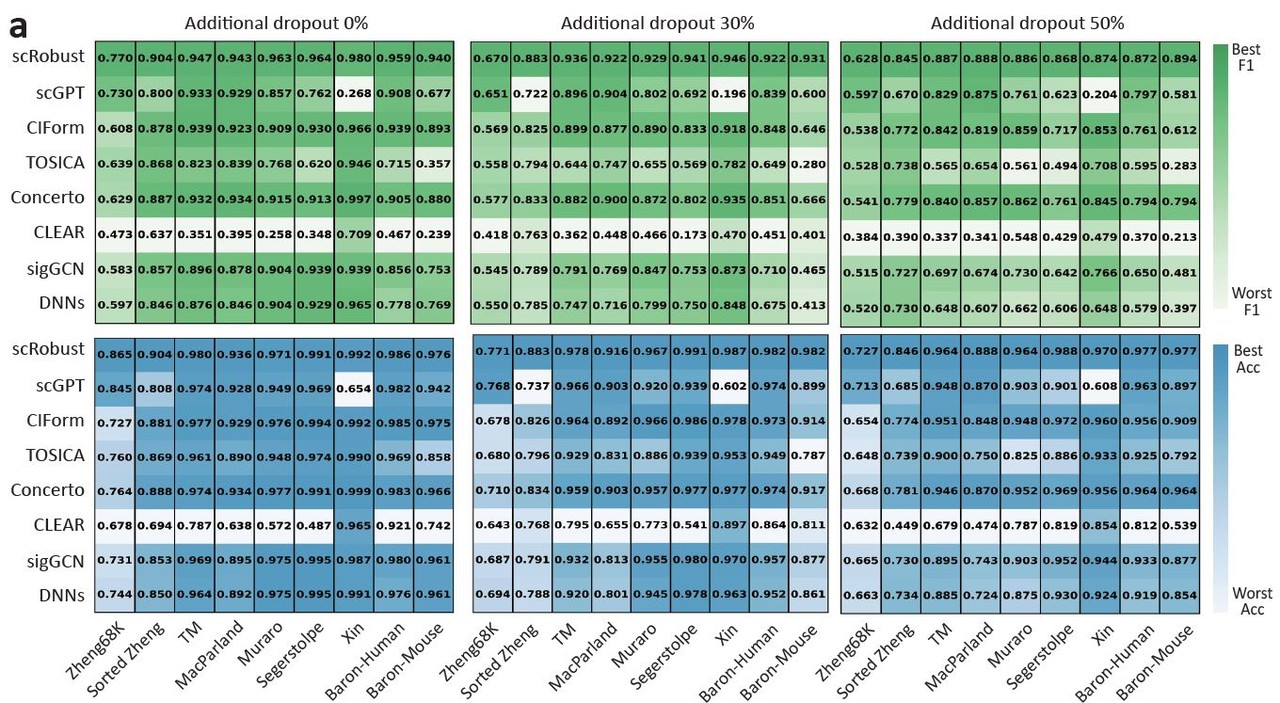
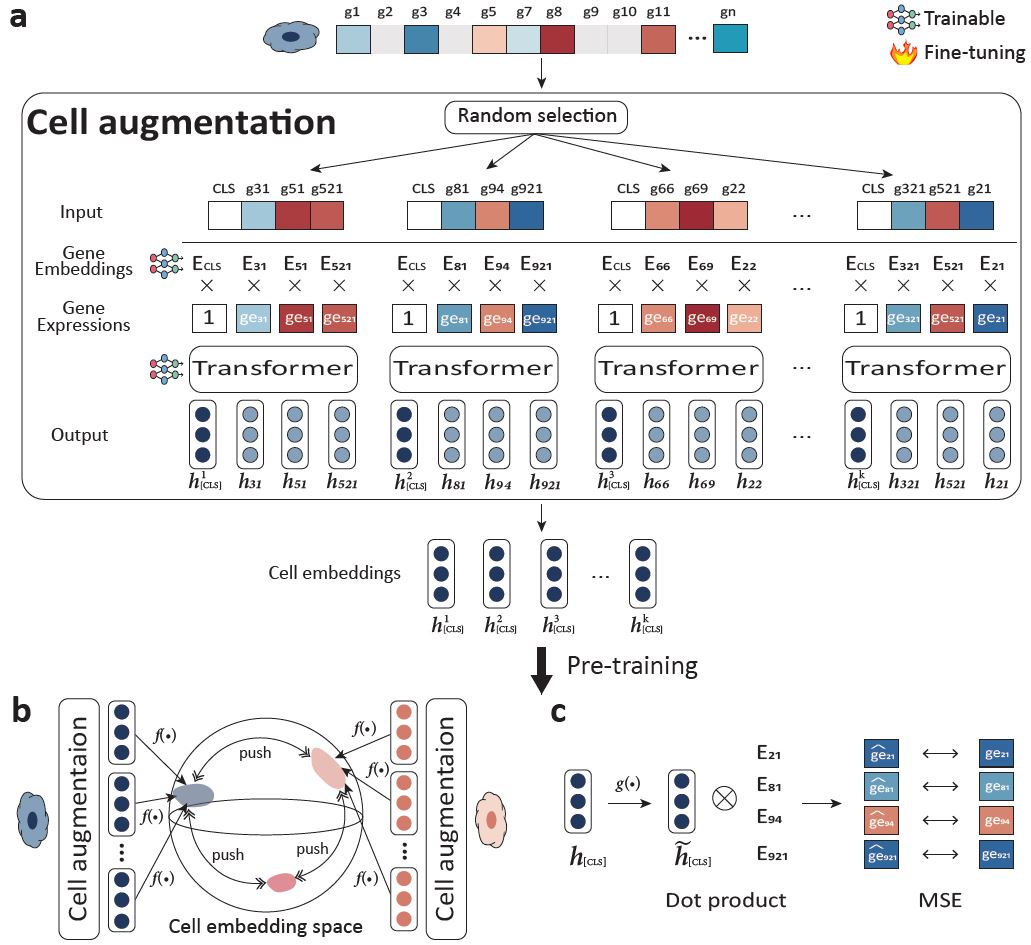
연구팀이 개발한 sc로버스트 방법론을 활용하면, 단 5% 미만의 유전자 정보만을 가지고도 각 단일세포의 보편적인 특징부터 세부적인 특징까지도 파악할 수 있게 된다.
하나의 세포로부터 다양한 유전자 조합을 만들어 여러 개의 세포 표현 벡터를 생성할 수 있는 방법론을 기반으로 단일세포 RNA 시퀀싱 데이터에 적합한 데이터 증강을 하는 것이다.
대조 학습을 통해 AI 모델을 학습시키면, 서로 다른 유전자 조합으로 생성된 세포 표현 벡터라도 같은 세포에서 나온 것인지, 다른 세포에서 나온 것인지 구분할 수 있다. 이 과정을 통해 다양한 유전자 조합으로 만든 세포 표현 벡터들이 하나의 통일된 세포 표현 벡터로 수렴하게 된다.
결과적으로 소수의 유전자만 사용하더라도 모든 유전자를 활용한 것과 유사한 세포 표현 벡터를 얻을 수 있어 전체 유전자를 사용하는 효과를 기대할 수 있다.

이현주 교수는 “이번 연구에서 개발된 알고리즘은 AI 모델이 유전자 일부만 학습하는 것이 아니라 모든 유전자에 대해 학습하는 것이 가능하다”라며 “이를 통해 그동안 소수의 세포에서만 발현되는 유전자와 같은 세포 유형에서 발생하는 미세한 특징들까지도 비교, 분석할 수 있게 되었다”라고 설명했다.
이어 “다양한 세포 종류의 마커 유전자뿐만 아니라 약물 저항성과 관련된 마커 유전자까지 추출할 수 있어 향후 단일세포 분석의 패러다임을 바꿀 수 있을 것으로 기대된다”라고 말했다.
이현주 GIST AI 대학원 교수가 지도하고 박세진 박사과정생이 수행한 이번 연구는 정보통신기획평가원(IITP)의 지원을 받았으며, 생물정보학 분야 JCR 상위 4% 국제학술지 'Briefings in Bioinformatics'에 2024년 11월16일 게재됐다.
박수빈 기자 sbin08@aitimes.com